ホーム > 原田 隆平/ Harada, Ryuhei
原田 隆平
Harada, Ryuhei
計算科学研究センター , 准教授 Center for Computational Sciences
-
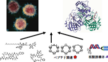
リボソーム内の化学的性質が翻訳中のタンパク質の立体構造に影響する
2024-08-20
原田 隆平

-
UBE学術振興財団第64回学術奨励賞
2024-04-01
原田 隆平

-
スーパーコンピュータを活用して「効く薬の候補」を素早く見つけ出す | 重田 育照
2021-04-01
重田 育照 広川 貴次 原田 隆平
オープンアクセス版の論文は「つくばリポジトリ」で読むことができます。
-
1.
Striatal Type I Interferon Responses Drive Disease Progression in the Amyotrophic Lateral Sclerosis
Shogo Tanabe, Keisuke Sekine, Koryo Obata, Terunori Sano, Akiko Uyeda, Lili Quan, Masaki Ueno, Ryuhei Harada, Koji Okamoto, Masaki Takao, Rieko Muramatsu
Science, submitted (2025)
-
2.
Chemical Diversity within Chaperonins Contributes to Stabilizing Substrate Protein Configurations
Takunori Yasuda; Yoshino Okamoto; Yasuteru Shigeta; Ryuhei Harada
The Journal of Physical Chemistry Letter, 16: 6321 (2025)
-
3.
Construction of PROTAC-Mediated Ternary Complex Structure Distribution Profiles Using Extensive Conformational Search
Genki Kudo; Takumi Hirao; Ryuhei Harada; Yasuteru Shigeta (+1 著者) Ryunosuke Yoshino
Journal of Chemical Information and Modeling 65: 6939 (2025)
-
4.
Rational Designed and Engineered Antimicrobial Peptides with High Selectivity and Efficiency Against Phytopathogenic Ralstonia Solanecearum
Melvalia Cristin Manik; Kikrusenuo Kiewhuo; Thanyada Rungrotmongkol; Lian Duan (+4 著者) Alisa Vangnai
Science of the Total Environment 976: 179354 (2025)
-
5.
In Silico Study on the Intrusion Process of an Inhibitor into AQP7 Channel Toward Efficient Drug Design
Koryo Obata; Yasuteru Shigeta; Ryuhei Harada
Chemistry Letters 54: upaf063 (2025)
-
6.
PaCS-Q: Enhanced Sampling Techniques in QM/MM Molecular Dynamics Simulations
Lian Duan; Kowit Hengphasatporn; Ryuhei Harada; Yasuteru Shigeta
Journal of Chemical Theory and Computation, 21: 4309 (2025)
-
7.
Plug-and-Play Toolkit for Ligand-Binding Path Sampling Parallel Cascade Selection Molecular Dynamics Simulation for AMBER and GROMACS
Ryuhei Harada; Kowit Hengphasatporn; Yasuteru Shigeta
(2025)
-
8.
Characterization of Delta-7 Alkenone Desaturase in Haptophytes Emiliania Huxleyi Through the Heterologous Expression in Tisochrysis Lutea
Kohei Yoneda; Chinatsu Kobayashi; Hiroya Araie; Rikuri Morita (+4 著者) Iwane Suzuki
Marine Biotechnology 27: 1 (2025)
-
9.
Unveiling the Antiviral Inhibitory Activity of Ebselen and Ebsulfur Derivatives on SARS-CoV-2 Using Machine Learning-based QSAR, LB-PaCS-MD, and Experimental Assay
Silpsiri Sinsulpsiri; Yuji Nishii; Qing-Feng Xu-Xu; Masahiro Miura (+12 著者) Thanyada Rungrotmongkol
Scientific Report 15: 6956 (2025)
-
10.
Phosphorylation on the N-terminal in Hmt1 Enhances Dimerization by Exposing the Binding Surface
Miyabi Endo; Takunori Yasuda; Rikuri Morita; Tomoaki Mizuno (+1 著者) Ryuhei Harada
Proteins: Structure, Function, and Bioinformatics, Manuscript Prot-00427-2024, under review: (2025)
-
11.
The Behavior of Water in Superheated Liquid Phase Observed in Molecular Dynamics Simulation
Morita Rikuri; Yasuteru Shigeta; Ryuhei Harada
Chemistry Letters, Manuscript ID CL-240416, under review: (2024)
-
12.
BEMM-GEN: A Tool for Generating a Biomolecular Environment Mimicking Model for Molecular Dynamics Simulation
Takunori Yasuda; Rikuri Morita; Yasuteru Shigeta; Ryuhei Harada
Journal of Chemical Information and Modeling 64: 7184 (2024)
-
13.
Structural Fluctuation in Homodimeric Aminoacyl-tRNA Synthetases Induces Half-of-the-sites Activity
Yoshino Okamoto; Takunori Yasuda; Rikuri Morita; Yasuteru ShigetaRyuhei Harada
The Journal of Physical Chemistry B 128: 10823 (2024)
-
14.
Evaluating Solubility, Stability, and Inclusion Complexation of Oxyresveratrol with Various β-cyclodextrin Derivatives Using Advanced Computational Techniques and Experimental Validation
Saba Ali; Aamir Aman; Kowit Hengphasatporn; Ryo Fujiki (+7 著者) Thanyada Rungrotmongkol
Computational Biology and Chemistry, 112: 108111 (2024)
-
15.
CommunicationMachine Learning-Based QSAR and LB-PaCS-MD Guided Design of SARS-CoV-2 Main Protease Inhibitors
Borwornlak Toopradab; Wanting Xie; Lian Duan; Kowit Hengphasatporn (+5 著者) Thanyada Rungrotmongkol
Bioorganic & Medicinal Chemistry Letters 110: 129852 (2024)
-
16.
Ribosome Tunnel Environment Drives the Formation of α-Helix During Co-Translational Folding
Takunori Yasuda; Rikuri Morita; Yasuteru Shigeta; Ryuhei Harada
Journal of Chemical Information and Modeling 64: 6610 (2024) Semantic Scholar
-
17.
Structural Generation by Inverse Transformation Using Principal Component Analysis Enhances Conformational Sampling of Protein
Rikuri Morita; Yasuteru Shigeta; Ryuhei Harada
Bulletin of the Chemical Society of Japan 97: uoae087 (2024)
-
18.
Prediction of the Binding Mechanism of a Selective DNA Methyltransferase 3A Inhibitor by Molecular Simulation
Genki Kudo; Takumi Hirao; Ryuhei Harada; Takatsugu Hirokawa (+1 著者) Ryunosuke Yoshino
Scientific Report 14: 13508 (2024)
-
19.
Alpha and Gamma Mangostins Inhibit Wild-type B SARS-CoV-2 More Effectively than the SARS-CoV-2 Variants and the Major Target is Unlikely a 3C-like Protease
Aphinya Suroengri; Van Cao; Patcharin Wilasluck; Peerapon Deetanya (+11 著者) Siwaporn Boonyasuppayakorn
Heliyon 10: e31987 (2024)
-
20.
Development of Membrane Permeability Coefficient by Means of Novel Molecular Dynamics Methods
Ryuhei Harada; Yuki Mitsuta; Yasuteru Shigeta
YAKUGAKU ZASSHI 144: 545 (2024)
書籍等出版物情報はまだありません。
-
41.
創薬研究を加速する分子シミュレーション技術の創出
原田 隆平
計算メディカルサイエンス 第1回全体ミーティング 2021年6月7日
-
42.
Developing a Modified Parameter for Evaluating Physical Property of Intrinsically Disordered Regions
Hayato Aida; Ryuhei Harada; Yasuteru Shigeta; Kentaro Tomii
第43回 日本分子生物学会 年会 2020年12月
-
43.
マルチウォーカーの再配置に基づく効率的なタンパク質構造サンプリング法の開発
保田 拓範; 森田 陸離; 重田 育照; 原田 隆平
第34回 分子シミュレーション討論会 2020年12月
-
44.
ld-PaCS-MD : PaCS-MDに基づくタンパク質−リガンド結合経路探索
會田 勇斗; 重田 育照; 原田 隆平
第34回 分子シミュレーション討論会 2020年12月
-
45.
モーフィングを利用してタンパク質の構造遷移を効率よく評価する
森田 陸離, 重田 育照, 原田 隆平
第34回 分子シミュレーション討論会 2020年12月
-
46.
PaCS-MDと異常検知を援用したレアイベントサンプリング法の開発
原田 隆平; 山口 孝太; 重田 育照
第34回 分子シミュレーション討論会 2020年12月
-
47.
A Development of Ligand Docking Parallel Cascade Selection Molecular Dynamics (ld-PaCS MD) and its Applications
Hayato Aida; Yasuteru Shigeta; Ryuhei Harada
第58回日本生物物理学会年会 2020年9月
-
48.
Theoretical Analyses on Dynamic Properties of DNA methyltransferase 1 and its Cofactors Based on All-atom Molecular Dynamics Simulations
Takunori Yasuda; Yasuteru Shigeta; Ryuhei Harada
第58回日本生物物理学会年会 2020年9月
-
49.
Development of de novo Method for Searching Protein-Ligands Binding Pathway: Ligand-Docking Parallel Cascade Selection Molecular Dynamics (ld-PaCS-MD)
Hayato Aida; Yasuteru Shigeta; Ryuhei Harada
2020 World Conference on Protein Science 2020年7月
-
50.
The Analyses of Dynamic Properties of Dnmt1 and Cofactors Based on All-atom Molecular Dynamics Simulations
Takunori Yasuda; Yasuteru Shigeta; Ryuhei Harada
2020 World Conference on Protein Science 2020年7月
-
51.
分散型シミュレーションに基づくレアイベントサンプリング法の開発
原田 隆平
化学反応のポテンシャル曲面とダイナミックス 2020年3月26日 招待有り
-
52.
新しい機能分子設計を目指した分子シミュレーション手法の開発
原田 隆平
第100回日本化学会春季年会「特別企画」分子のレジデンスを考える−新しい機能分子設計の鍵として− 2020年3月22日 招待有り
-
53.
生体機能を解明する分子シミュレーション手法の開発と応用
原田 隆平
理論タンパク質物性科学の最前線:理論と実験との密な協同 2020年2月27日 招待有り
-
54.
タンパク質の機能発現に重要なレアイベ ントを抽出するサンプリング手法の開発 (2019年度 分子シミュレーション学会 学術賞 受賞講演)
原田 隆平
第33回分子シミュレーション討論会 2019年12月10日 招待有り
-
55.
中分子環状ペプチドの膜透過性を評価する分子シミュレーション手法の開発
原田 隆平
計算メディカルサイエンス事業部発足キックオフシンポジウム 2019年12月6日 招待有り
-
56.
カスケード選択型分子動力学シミュレーションで実現する環状ペプチドの膜透過シミュレーション
原田 隆平
東京大学 情報基盤センター H31 インターン・前期「若手・女性利用者推薦」成果報告会 2019年12月3日 招待有り
-
57.
レアイベントサンプリング法の開発と応用研究
原田 隆平; Sladek Vladimir; 重田 育照
日本コンピュータ化学会2019年秋季年会 2019年10月24日
-
58.
生体機能を解明する分子シミュレーション手法の開発と応用
原田 隆平
筑波大学 プレ戦略研究会「生命から学ぶ」 2019年10月10日 招待有り
-
59.
in slicoタンパク質設計を実現する分子シミュレーション手法の構築
原田 隆平
生物工学会シンポジウム"in silicoタンパク質設計で加速するタンパク質工学・応用構造生物学" 2019年9月18日 招待有り
-
60.
データ駆動型分子動力学シミュレーションに立脚した効率的タンパク質構造サンプリング手法の開発
原田 隆平
第408回CBI学会講演会 「分子シミュレーション技術の新たなる展望〜AI活用, 高精度力場, データ駆動型シミュレーション」 2019年7月16日 招待有り
-
1. 特願2015-156703: 情報処理装置, 指標次元抽出方法, および指標次元抽出プログラム
中村 朋健; 原田 隆平; 重田 育照 -
2. 特願15/228,873: Information Processing Apparatus and Index Dimension Extracting Method
Nakamura Tomotake; Harada Ryuhei; Shigeta Yasuteru
2,432 total views